7.4 Poisson regressie
Poissonregressie werkt hetzelfde als binomiale, maar je gebruikt family = poisson.
Zo simpel is dat.
Voorbeeld van data over aantal overreden amfibiën t.o.v. afstand tot een Nationaal Park in Portugal:
roadkill %>%
ggplot(aes(D.PARK, TOT.N)) +
geom_point()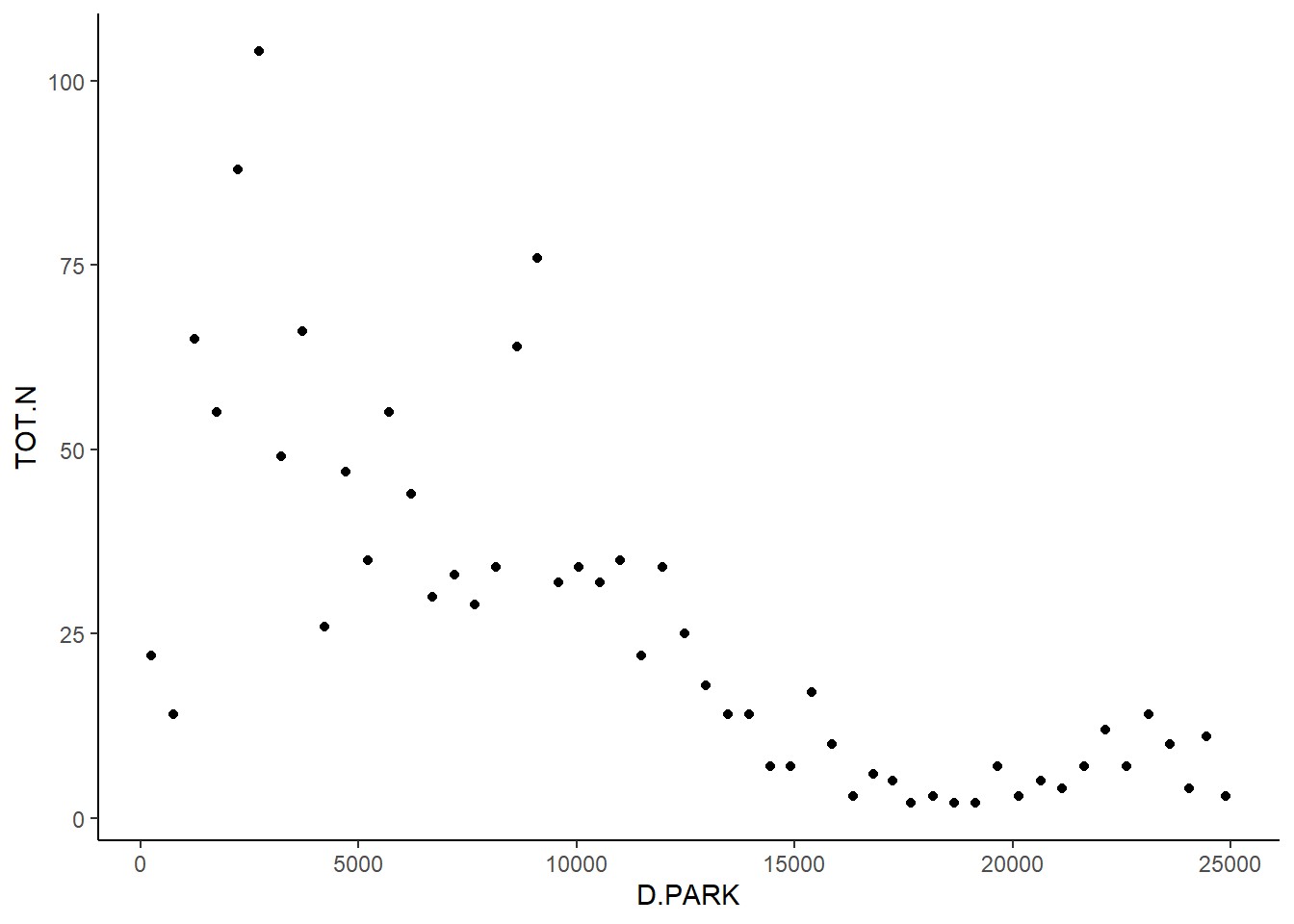
Hier kunnen we een regressie op loslaten:
fit <- glm(TOT.N ~ D.PARK, data = roadkill, family = poisson)
summary(fit)##
## Call:
## glm(formula = TOT.N ~ D.PARK, family = poisson, data = roadkill)
##
## Deviance Residuals:
## Min 1Q Median 3Q Max
## -8.1100 -1.6950 -0.4708 1.4206 7.3337
##
## Coefficients:
## Estimate Std. Error z value Pr(>|z|)
## (Intercept) 4.316e+00 4.322e-02 99.87 <2e-16 ***
## D.PARK -1.059e-04 4.387e-06 -24.13 <2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## (Dispersion parameter for poisson family taken to be 1)
##
## Null deviance: 1071.4 on 51 degrees of freedom
## Residual deviance: 390.9 on 50 degrees of freedom
## AIC: 634.29
##
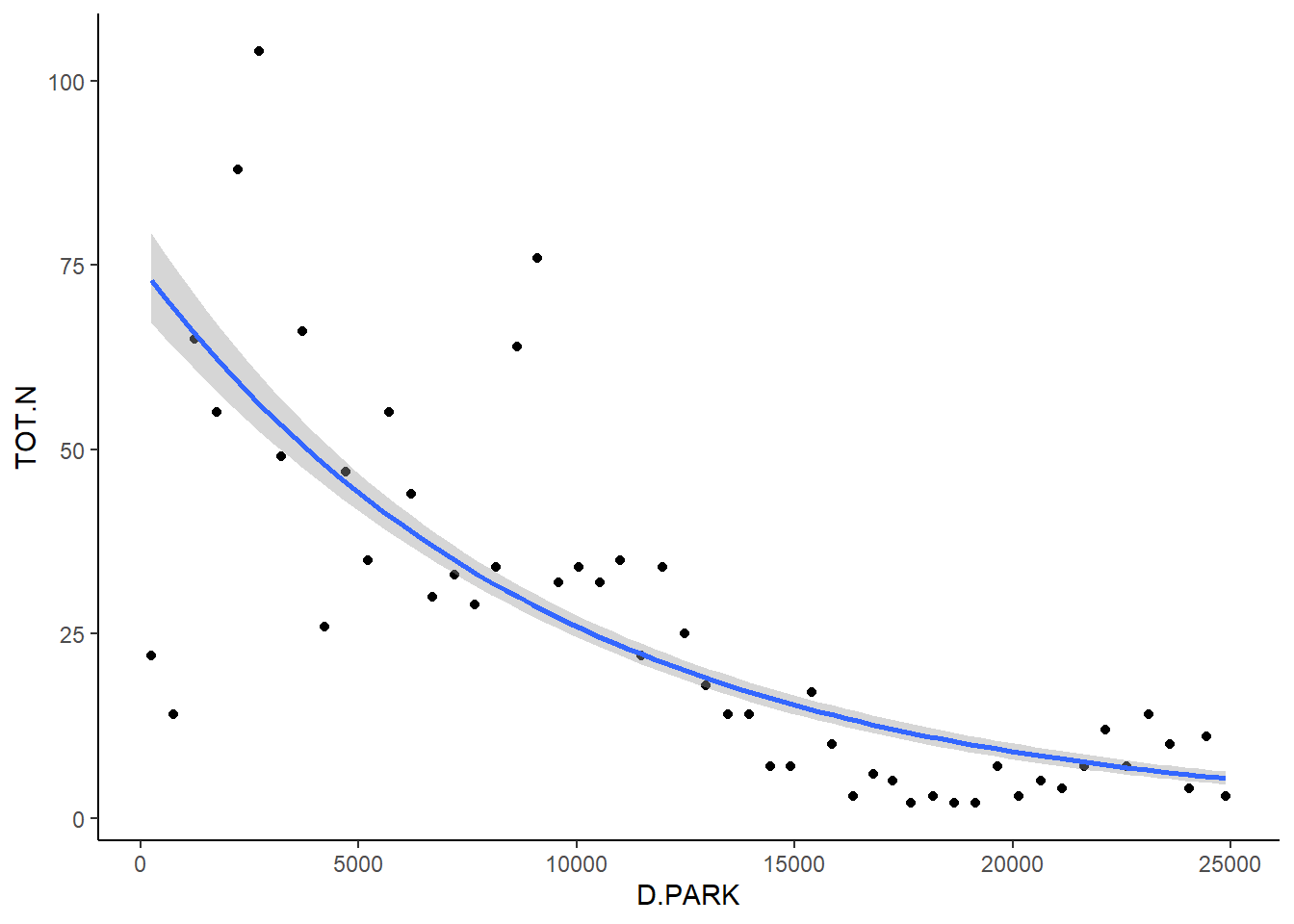
## Number of Fisher Scoring iterations: 4Met functie geom_smooth kan je ook zulk soort modellen fitten op je data in een grafiek:
roadkill %>%
ggplot(aes(D.PARK, TOT.N)) +
geom_point() +
geom_smooth(method = 'glm', method.args = list(family = poisson))