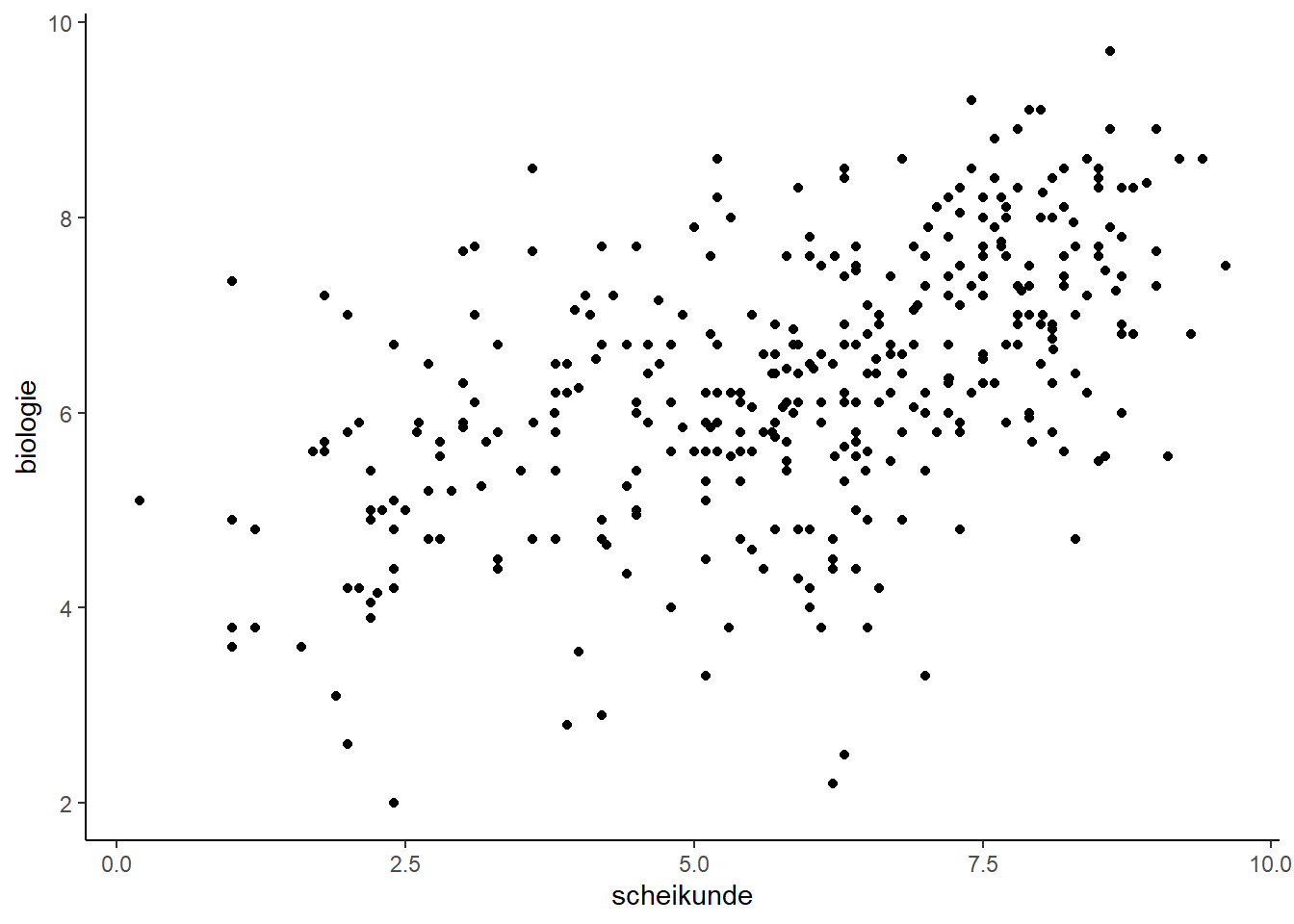
2.1 Scatterplot
- Package tidyverse laden
- dataframe cijfers in pipeline laden
- figuur opzetten met op x-as scheikunde en y-as biologie (uit dataframe cijfers)
- Scatterplot toevoegen
- Thema veranderen naar classic
NB: je kan een thema permament instellen (die dus geldt voor iedere figuur die je maakt in die sessie):
theme_set(theme_classic())2.1.1 Scatterplot met regressielijn
- Deze regel voegt een regressielijn toe (op basis van een lineair model (lm)).
## `geom_smooth()` using formula 'y ~ x'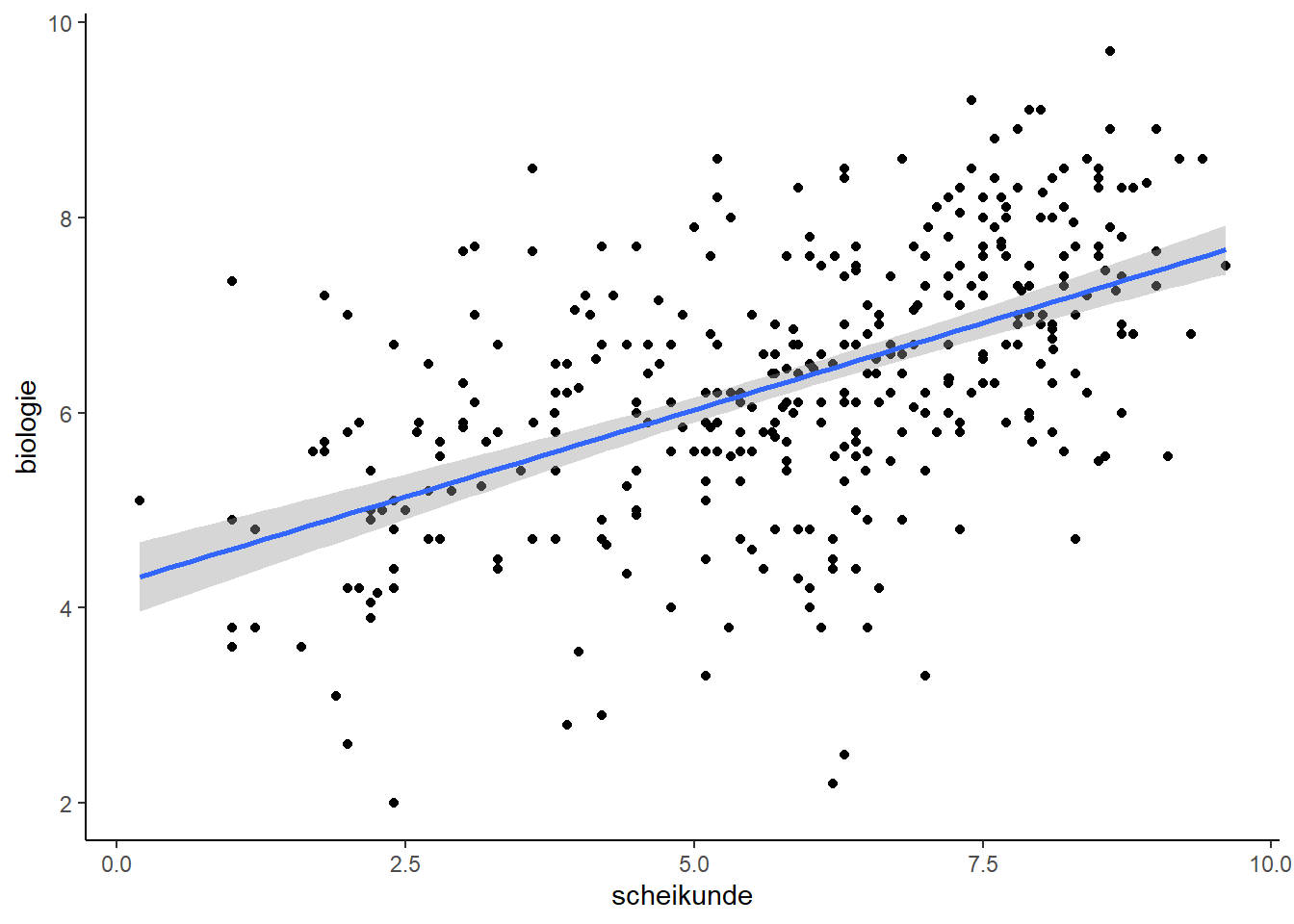
Wil je geen betrouwbaarheidsinterval rondom de lijn: geom_smooth(method="lm", se=FALSE)
2.1.2 1:1-lijn
Wil je een zogenaamde 1:1-lijn, om te checken of de studenten stelselmatig een hoger cijfer voor biologie:
- Voegt een lijn toe waarbij je richtingscoëfficient (slope) en/of snijpunt y-as (intercept) kiest.
cijfers %>%
ggplot(aes(scheikunde, biologie)) +
geom_point() +
geom_abline(slope = 1) +
theme_classic()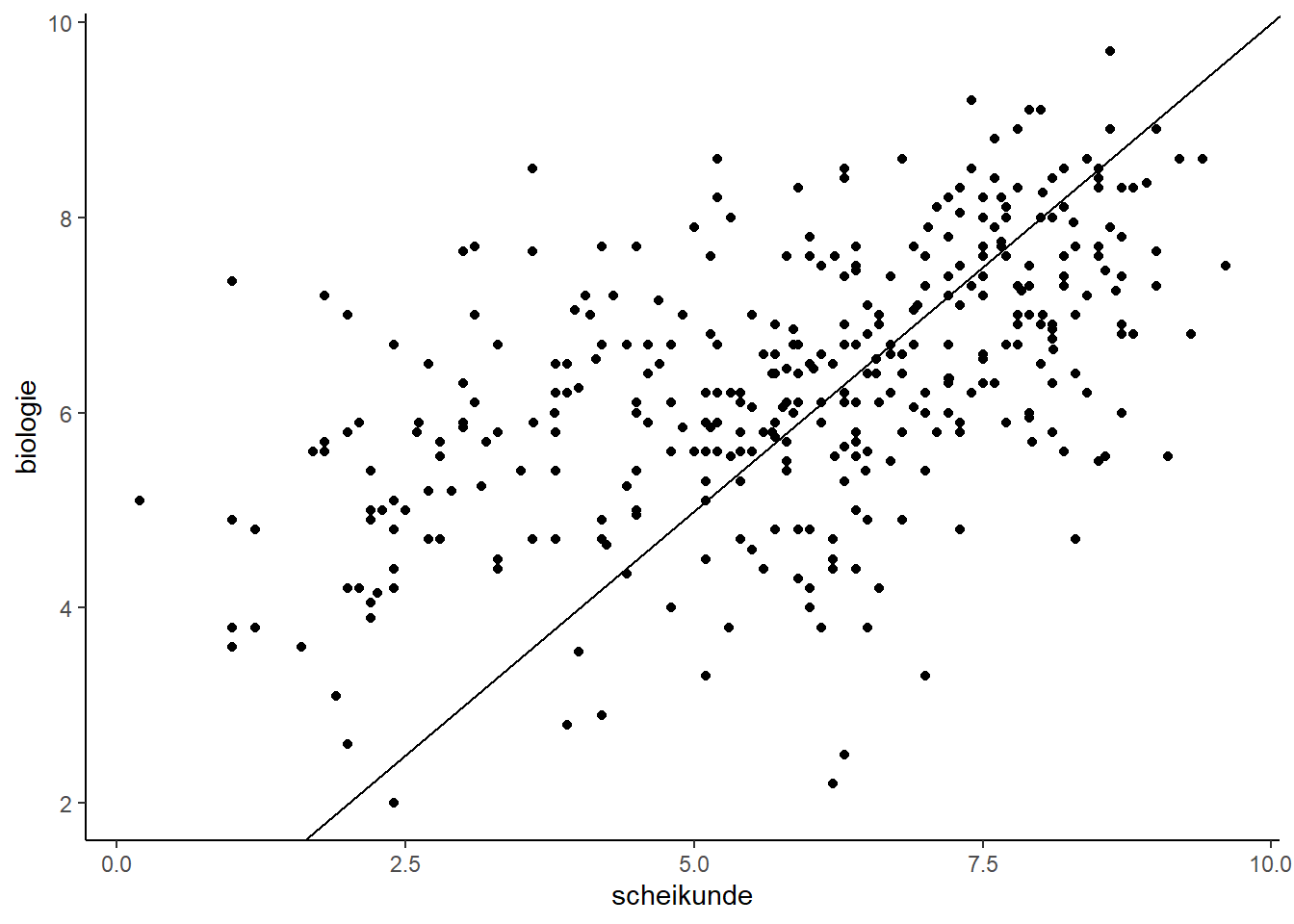
2.1.3 Limieten assen aanpassen
- met deze regel verander je limiet x-as
- met deze regel verander je limiet y-as
cijfers %>%
ggplot(aes(scheikunde, biologie)) +
geom_point() +
geom_abline(slope = 1) +
xlim(1, 10) +
ylim(1, 10)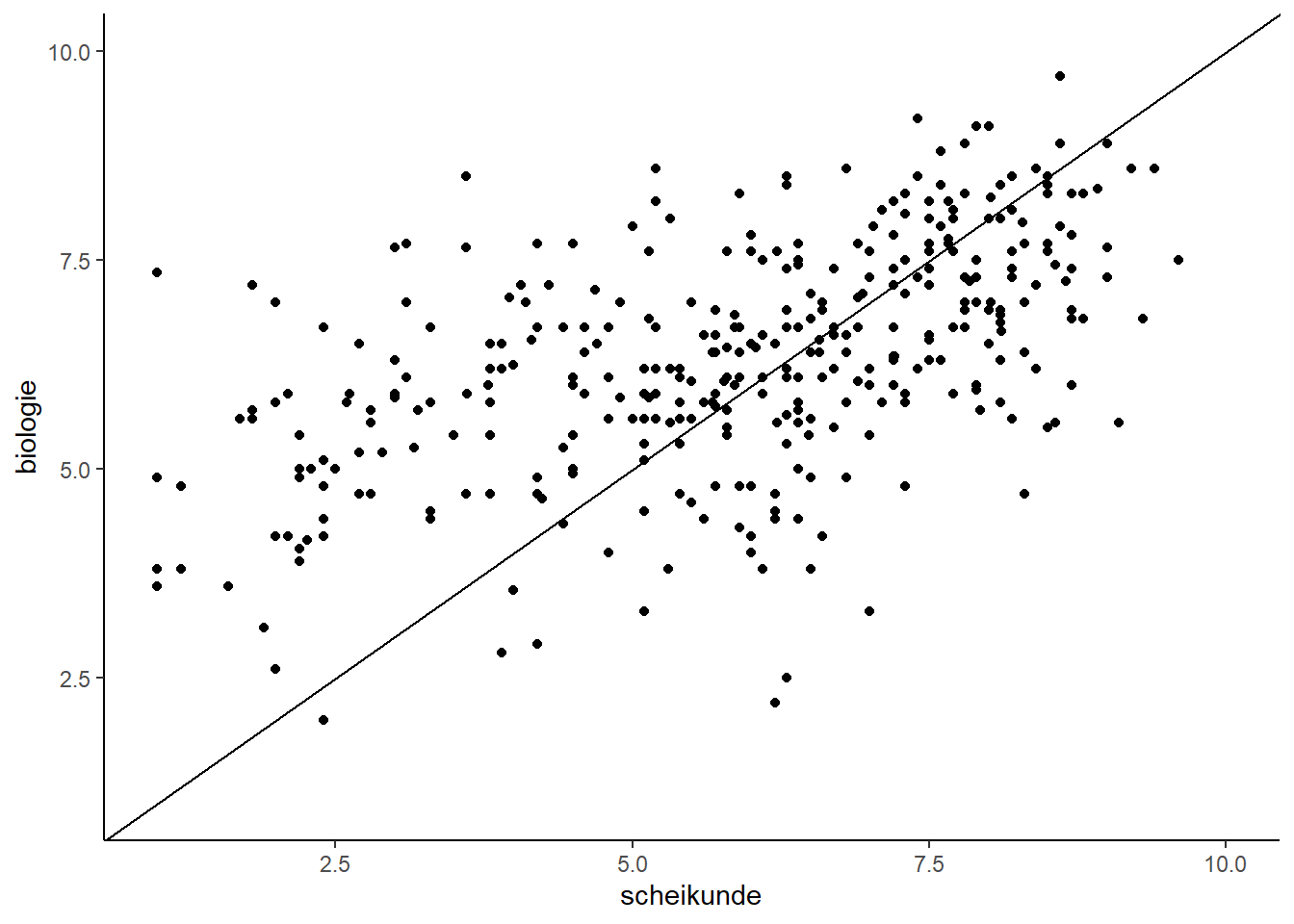
2.1.4 Data in groepen verdelen
- Met argument colour of fill kan je je data in groepen verdelen op basis van een variabele (in dit geval geslacht).
cijfers %>%
ggplot(aes(scheikunde, biologie, colour = geslacht)) +
geom_point() +
geom_abline(slope = 1) +
xlim(1, 10) +
ylim(1, 10)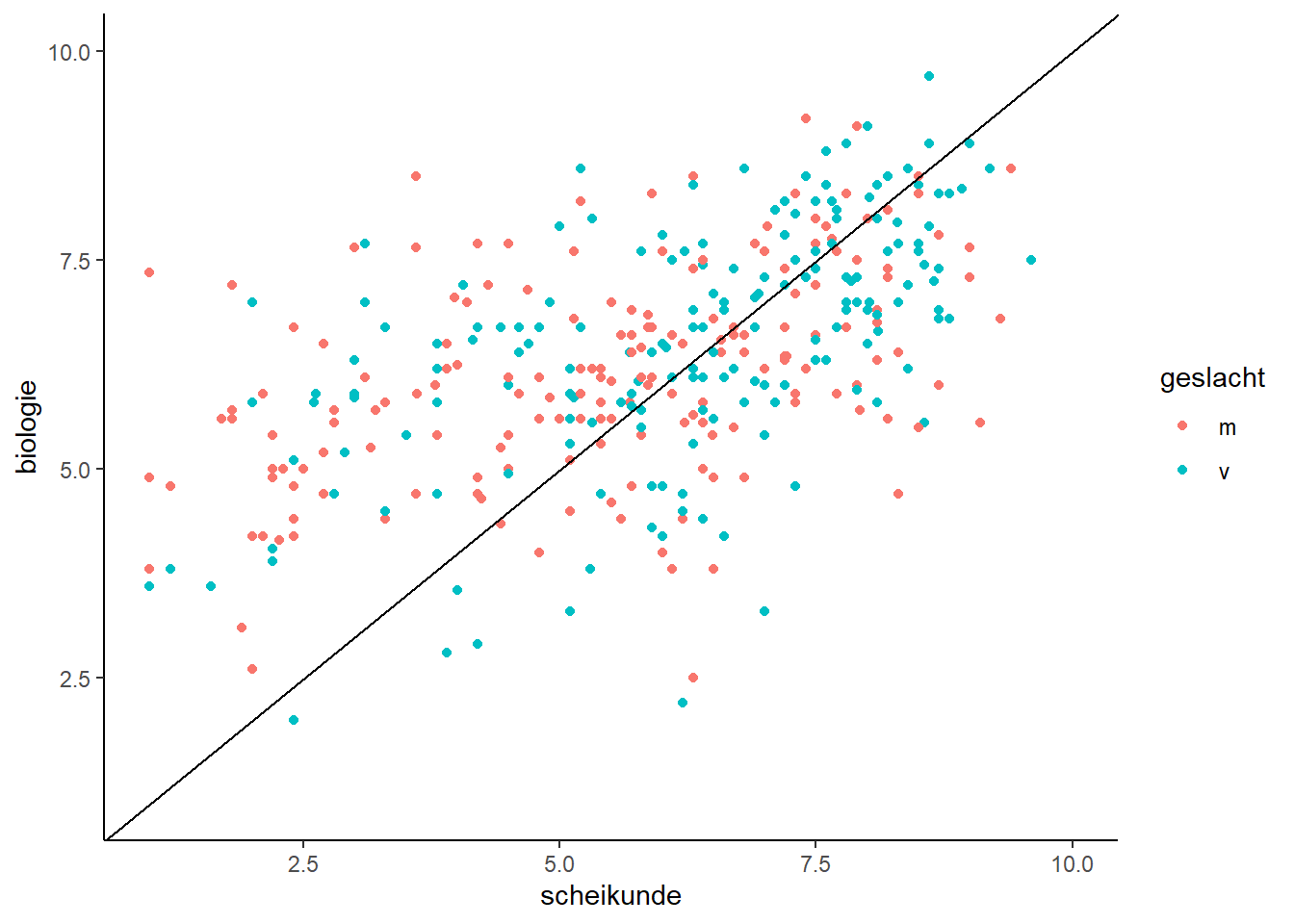
2.1.5 Handmatig kleur per groep bepalen
- Met deze regel kan je handmatig kleuren toekennen aan groepen. Laat je deze regel weg, dan kiest ggplot zelf zijn kleuren.
cijfers %>%
ggplot(aes(scheikunde, biologie, colour = geslacht)) +
scale_color_manual(values = c("m"="blue", "v"="deeppink1")) +
geom_point() +
geom_abline(slope = 1) +
xlim(1, 10) +
ylim(1, 10)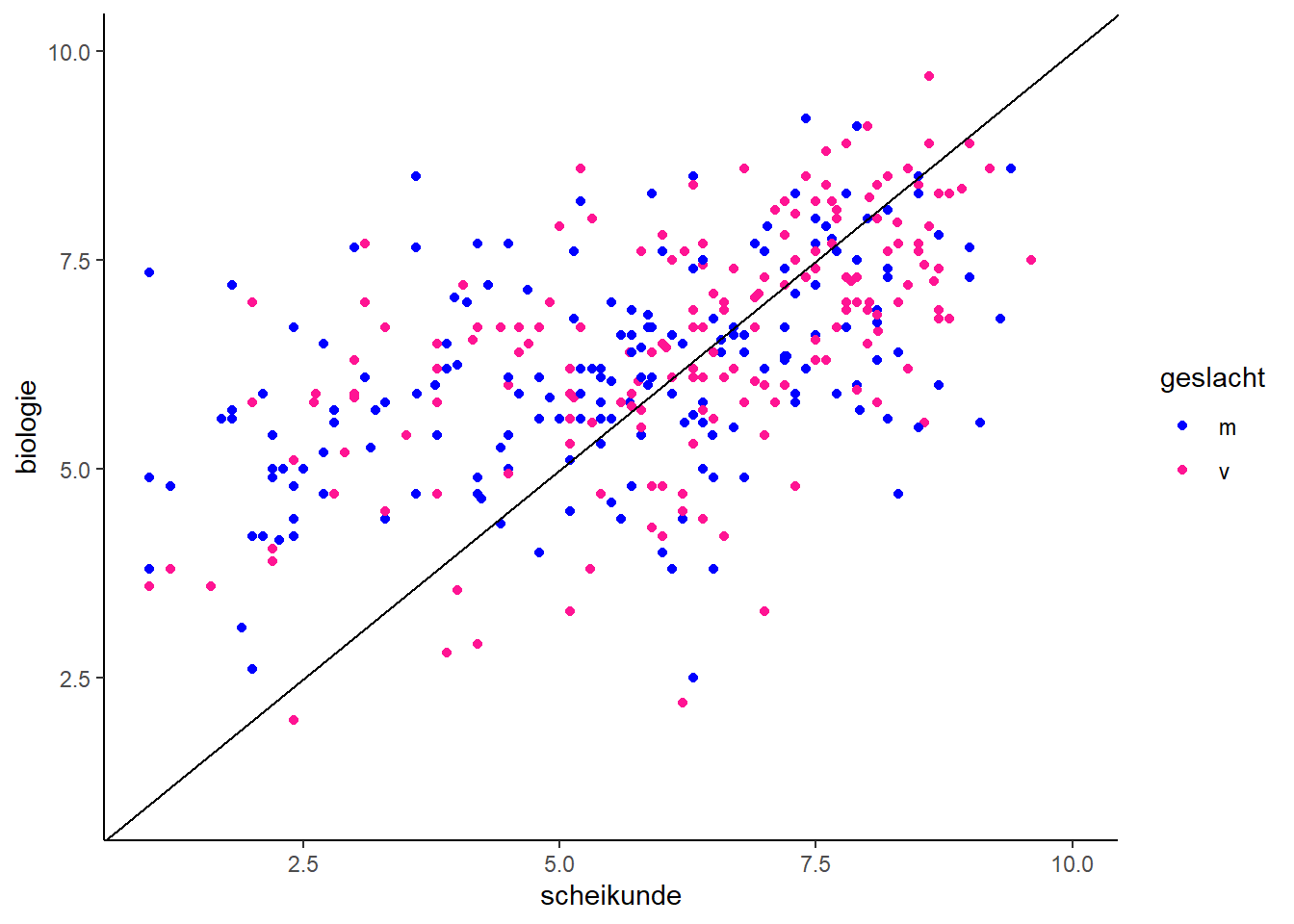
2.1.6 Per groep symbool toekennen
- Met deze regel geef je per groep aan met wat voor symbool het weergegeven wordt. Je kan kiezen uit een aantal basisvormen (opgeroepen met cijfers 1 t/m 25), of bijv. unicode zoals hier.
cijfers %>%
ggplot(aes(scheikunde, biologie, shape=geslacht,
colour=geslacht)) +
scale_shape_manual(values = c("m"="\u2642", "v"="\u2640")) +
scale_color_manual(values = c("m"="blue", "v"="deeppink1")) +
geom_point(size=2) +
geom_abline(slope=1) +
xlim(1,10) +
ylim(1,10)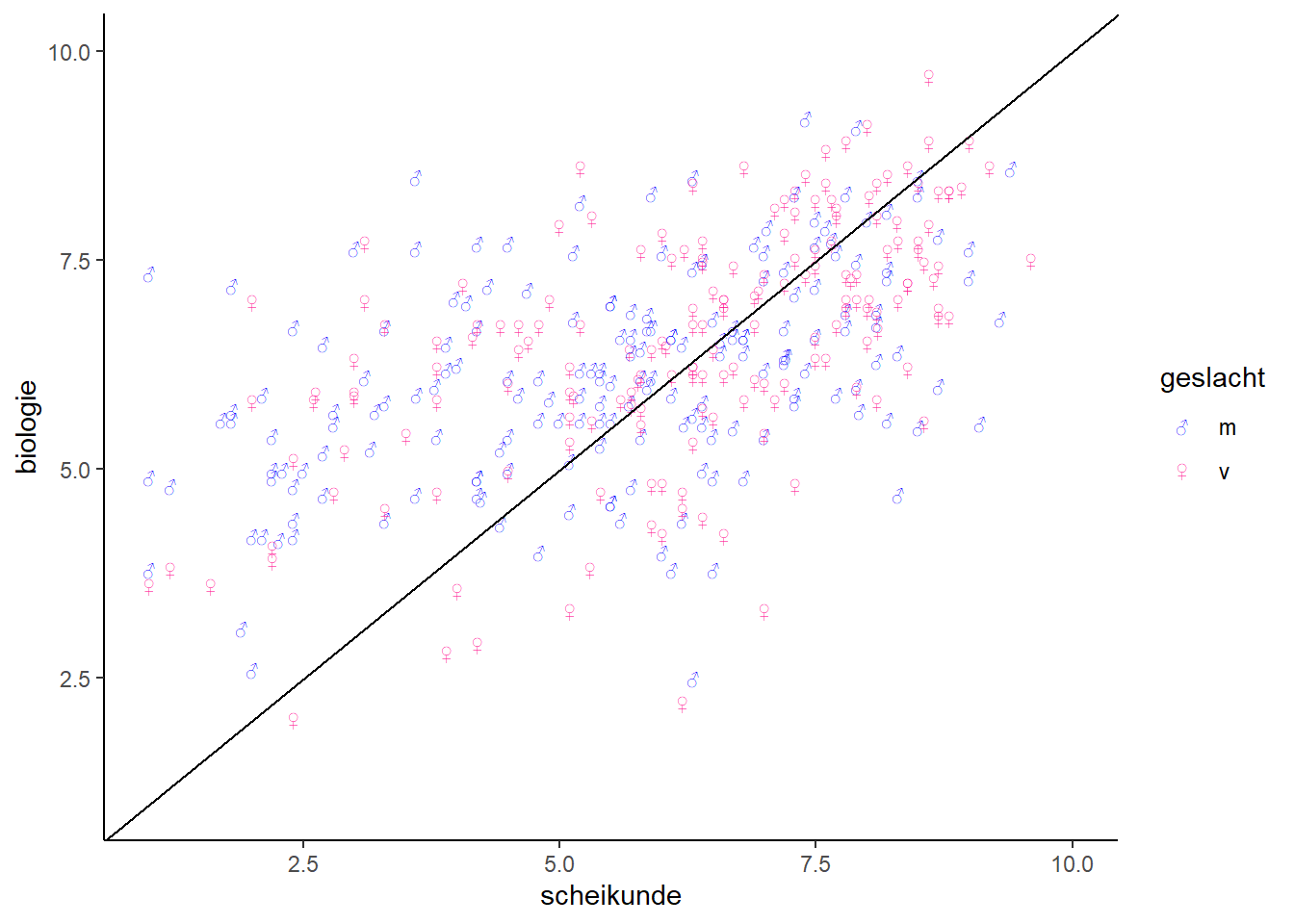
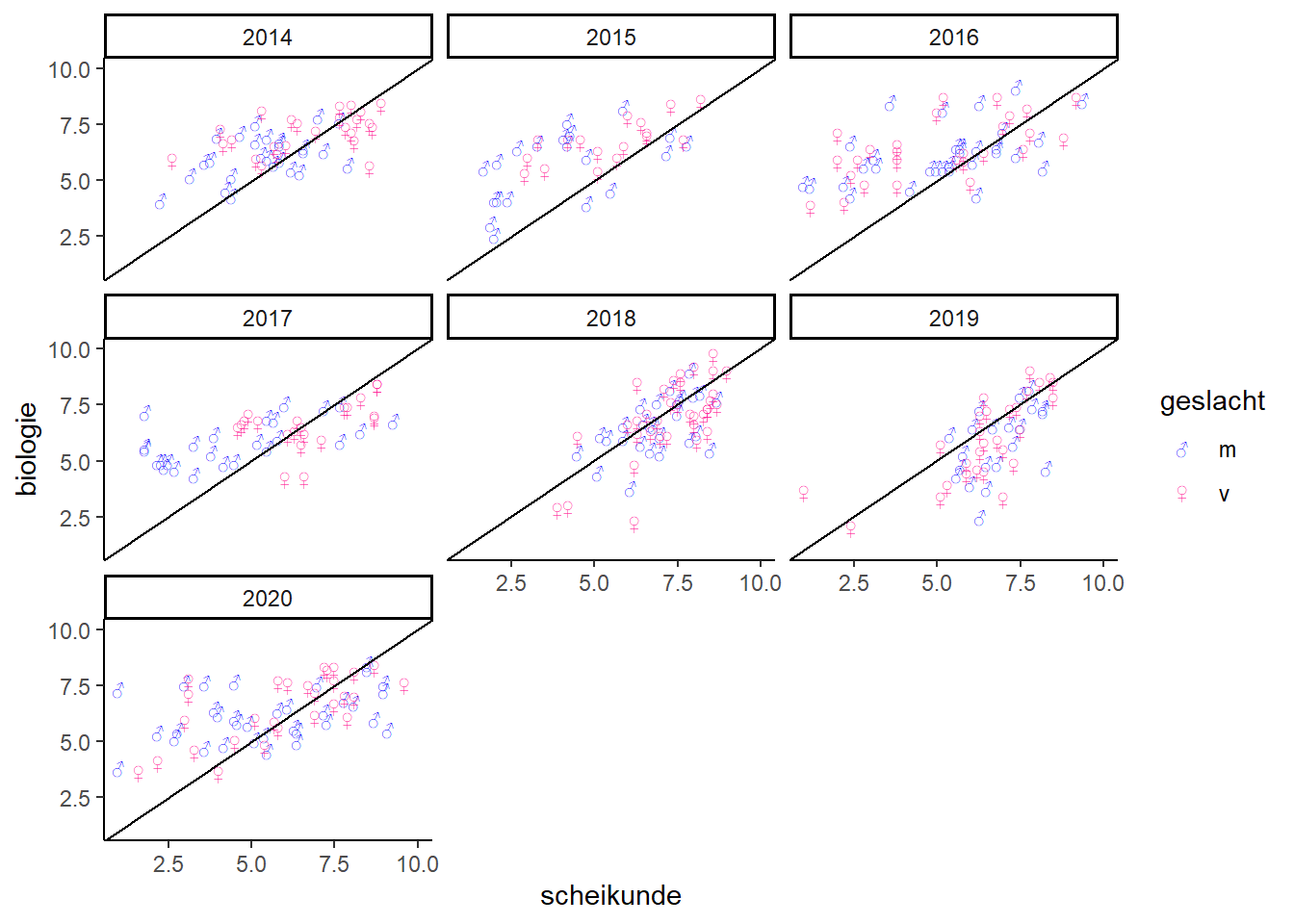
2.1.7 Data verdelen in meerdere grafieken
De cijferdata omvat vijf jaargangen eerstejaarsstudenten.
- Met facet_wrap verdeel je de data in meerdere figuren (in dit geval op basis van jaar)
cijfers %>%
ggplot(aes(scheikunde, biologie, shape=geslacht,
colour=geslacht)) +
scale_shape_manual(values = c("m"="\u2642", "v"="\u2640")) +
scale_color_manual(values = c("m"="blue", "v"="deeppink1")) +
geom_point(size=2) +
geom_abline(slope=1) +
facet_wrap(vars(jaar)) +
xlim(1,10) +
ylim(1,10)